Resumo
O linfoma de Hodgkin clássico (cHL) é geralmente caracterizado por um baixo conteúdo de células tumorais, derivadas de células B do centro germinativo aleijadas. Raros casos foram descritos em que as células tumorais mostram rearranjos clonais do receptor de células T. Do ponto de vista clínico-patológico, não está claro se esses casos devem ser classificados como cHL ou linfoma anaplásico de grandes células T (ALCL). Uma vez que observamos recentemente diferenças na motilidade de células tumorais ALCL e cHL, aqui, pretendemos obter uma melhor compreensão do cHL derivado de células T, investigando seus perfis proteômicos globais e sua motilidade. Em uma análise proteômica, quando apenas proteínas associadas à motilidade foram consideradas, as linhagens de células cHL derivadas de células T mostraram a maior semelhança com ALK −Linhas celulares ALCL. Em contraste, as linhagens de células cHL derivadas de células T apresentaram uma motilidade global muito baixa, semelhante à observada em cHL convencional. Enquanto todas as linhagens de células ALCL, bem como cHL derivada de células T, apresentaram predominantemente um padrão de migração amebóide com urópodes na parte posterior, cHL convencional nunca apresentou urópodes. A migração de linhagens celulares ALCL foi fortemente prejudicada pela aplicação de diferentes inibidores. Este efeito foi menos pronunciado em linhas de células cHL e quase invisível em cHL derivado de células T. Em resumo, nossos dados derivados de linhagens celulares sugerem que, com base na proteômica e no comportamento de migração, o cHL derivado de células T é uma neoplasia que compartilha características com o cHL e o ALCL e não é um ALCL com baixo conteúdo de células tumorais. Estudos clínicos complementares sobre este linfoma são necessários.
1. INTRODUÇÃO
O linfoma de Hodgkin clássico (cHL) e o linfoma anaplásico de grandes células (ALCL) representam linfomas com células tumorais CD30+. 1 cHL é frequentemente diagnosticado em estágios iniciais da doença, 2 enquanto ALCL é mais frequentemente encontrado em estágio avançado da doença e tem um resultado menos favorável. 3 Apesar de esses linfomas se originarem de células fundamentalmente diferentes, com origem em células B para cHL 4 e origem em células T para ALCL, 5 ambos os linfomas têm várias características em comum: ambos perderam a expressão de marcadores de superfície específicos de linhagem e ambos têm sinalização NFKappaB, NOTCH1, AP1 e JAK-STAT constitutivamente ativa. 6 , 7 ALCL é classificado em ALK+ ALCL, com rearranjo do gene ALK, e ALK − ALCL, que pode abrigar rearranjos de DUSP22 ou TP63 . 8 , 9 Para ALK + ALCL, é descrito um subtipo do tipo Hodgkin, que é particularmente difícil de diferenciar do cHL puramente pela morfologia. 10 Nesse caso, a expressão da proteína ALK1 é uma ferramenta diagnóstica útil. No entanto, ALK − ALCL também pode apresentar uma baixa carga tumoral e, portanto, pode ser difícil de distinguir de cHL, uma vez que ALK1 não é expresso por ALK −ALCL. Normalmente, a demonstração da coloração nuclear do PAX5 é muito útil nesse cenário, pois o PAX5 geralmente é expresso em cHL, mas não em ALCL. 11 No entanto, a classificação de alguns casos que não expressam PAX5 permanece incerta. PAX5 − casos de cHL mostraram ter sobrevida inferior em comparação com os casos convencionais de cHL. 12 A demonstração de rearranjos clonais de receptores de células B ou T (TCR) pode ser útil em alguns casos, mas devido à baixa carga de células tumorais e à extensa hipermutação somática de genes de imunoglobulina no cHL, os rearranjos clonais são frequentemente perdidos. A coloração imuno-histoquímica pode fornecer alguma ajuda no diagnóstico. 13Em um subconjunto de casos, foi observada uma associação com ALCL cutâneo, com células tumorais CD30 + únicas, espalhadas e grandes nos linfonodos que mimetizam cHL. 14 Esses casos representam uma manifestação nodal de um ALCL cutâneo. No entanto, poucos casos permanecem, em que nenhuma ligação com um linfoma cutâneo de células T pode ser demonstrada. Com base na reação em cadeia da polimerase unicelular (PCR), rearranjos clonais do TCR podem ser demonstrados nesses casos. 15 No entanto, mesmo duas das linhagens de células cHL reconhecidas, a saber, L-540 e HDLM-2, abrigam rearranjos clonais de TCR, originando-se assim de células T. O HDLM-2 foi estabelecido a partir do derrame pleural de um homem de 74 anos com linfoma de Hodgkin (esclerosante nodular; estágio IV) em 1982. 16L-540 foi estabelecido a partir da medula óssea de uma mulher de 20 anos com linfoma de Hodgkin (esclerosante nodular; estágio IVB). 17 Em estudos de expressão gênica, essas linhagens celulares se agruparam muito bem com cHL. 18 Assim, a biologia dos casos com morfologia de cHL e rearranjo clonal de TCR nas células tumorais ainda não está clara. A morfologia típica e o imunofenótipo das células tumorais, o microambiente e os genes do receptor de células B aleijados fazem parte da definição de cHL. No entanto, nestes casos particulares com morfologia de cHL e rearranjo clonal de TCR nas células neoplásicas, os critérios tradicionais de classificação não ajudam mais. Como observamos recentemente que os linfomas malignos diferem em suas propriedades migratórias, 19o presente estudo partiu da ideia de que uma análise da motilidade dessas células tumorais poderia ajudar a compreender e classificar melhor esses linfomas. Portanto, o presente estudo teve como objetivo entender melhor a biologia e classificação das neoplasias de cHL derivadas de células T com foco nas linhagens de células cHL derivadas de células T L-540 e HDLM-2 e seu comportamento migratório.
2 MÉTODOS
2.1 Proteômica
As linhas celulares L-428 ( RRID:CVCL_1361 ), L-1236 ( RRID:CVCL_2096 ), DEL ( RRID:CVCL_1170 ), SU-DHL-1 (CVCL_0538), HDLM-2 ( RRID:CVCL_0009 ) e L-540 ( RRID:CVCL_1362 ) foram adquiridos da German Collection of Microorganisms and Cell Cultures (Braunschweig, Alemanha). As linhas celulares Mac-1 ( RRID:CVCL_H631 ) e Mac-2A ( RRID:CVCL_H637 ) foram fornecidas pelo Prof. Olaf Merkel, Universidade Médica de Viena, Áustria. A identidade de todas as linhas celulares foi autenticada por perfis de repetições curtas em tandem (STR). Todas as linhagens celulares foram testadas regularmente quanto à contaminação por micoplasma.
Todas as linhas celulares foram submetidas a proteômica quantitativa com base em marcação de massa em tandem (TMT). Para isso, 5 x 10 5 células foram colhidas e lisadas em tampão de ureia como descrito anteriormente. 20
Após extração proteica e digestão com tripsina, os peptídeos foram marcados com reagentes TMT 10-plex (Thermo Fisher Scientific, Dreieich, Alemanha). Os peptídeos marcados individualmente foram combinados em amostras multiplexadas. Para normalização entre multiplexes, foi incluída uma referência interna composta por peptídeos de cada condição. Após pré-fracionamento usando um kit de fase reversa C18 de pH alto (Thermo Fisher Scientific), as misturas de peptídeos marcados com TMT foram separadas por HPLC de fase reversa C18 em um sistema Ultimate 3000 RSLCnano (Thermo Fisher Scientific). Os peptídeos eluídos da coluna analítica foram analisados por espectrometria de massa nano-ESI Orbitrap em tandem em um instrumento Q Exactive HF (Thermo Fisher Scientific). Os dados de espectrometria de massa bruta foram processados com o software MaxQuant (versão 1.6.17.0;21 Isso incluiu a identificação de proteínas executando os espectros de massa contra o banco de dados de proteínas humanas Uniprot (baixado em fevereiro de 2019) e coleta de contaminantes laboratoriais comuns, seguido pela extração de intensidades de íons repórter TMT para quantificação de proteínas. Após a recalibração, as tolerâncias de massa para íons precursores e fragmentos foram ajustadas para 4,5 e 20 ppm, respectivamente. Oxidação da metionina e acetilação da proteínaN-terminais foram consideradas modificações variáveis, e a carbamidometilação da cisteína foi definida como uma modificação fixa. O comprimento mínimo do peptídeo foi ajustado para sete aminoácidos, permitindo até duas clivagens trípticas perdidas. Tanto nos níveis de peptídeo quanto de proteína, uma taxa máxima de falsa descoberta (FDR) de 1% foi aplicada usando um banco de dados de isca reversa. Para processamento de dados a jusante com o software Perseus (versão 1.6.0.7; MPIB), foram removidos contaminantes potenciais, proteínas identificadas apenas com peptídeos modificados e acertos no banco de dados chamariz. Após a normalização das intensidades do íon repórter TMT em amostras multiplexadas, os dados foram submetidos a agrupamento hierárquico não supervisionado com base na distância euclidiana e no método de ligação média, usando todos os grupos de proteínas quantificados ou após o mapeamento de proteínas relacionadas à motilidade.22 repositório de parceiros com o identificador de conjunto de dados PXD031907 (https://www.ebi.ac.uk/pride).
2.2 Experimentos de microcanais
Chips de polidimetilsiloxano (PDMS) com diferentes tipos de microcanais foram produzidos em moldes fornecidos pelo Dr. Matthieu Piel. Canais retos com um diâmetro de 8 µm e altura de 10 µm foram testados e considerados mais apropriados para as células tumorais bastante grandes de Hodgkin e Reed-Sternberg (HRS) de linhagens celulares cHL. Para experimentos de constrição, foram escolhidos canais com formato de 12 µm × 12 µm e constrição de 12 µm × 4 µm. Os microcanais foram revestidos com fibronectina por pelo menos 1 h. Os inibidores Y27632 (Meck, Darmstadt, Alemanha), blebbistatina e CK-666 (ambos Cayman Chemicals, Ann Arbor, MI, EUA) foram aplicados em doses especificadas antes das células serem carregadas nos chips. Antes dos experimentos, as doses ideais de inibidor foram determinadas por uma curva de titulação, identificando dosagens que tiveram apenas efeitos menores na viabilidade celular (dados não mostrados).5 células com ou sem inibidores foram carregadas no chip PDMS e o chip foi pré-incubado por 1 h na incubadora. A motilidade celular foi monitorada usando um microscópio de contraste de fase em condições de incubadora por 20 h, com imagens de lapso de tempo tomadas a cada 10 min em canais retos e a cada 4 min em microcanais com constrições. As células em vídeos de experimentos de microcanal foram segmentadas com um script personalizado (disponível mediante solicitação; veja abaixo uma descrição do procedimento). As posições X e Y das células segmentadas foram usadas como entrada para rastreamento, que foi feito com o TrackMate 23usando o rastreador LAP (parâmetros: distância máxima para vinculação quadro a quadro: 100; distância máxima para fechamento de lacuna: 150; comprimento máximo de lacuna: 15; penalidade para vincular células com diferentes posições Y: Y15). A velocidade e a retilineidade com base no degrau foram calculadas com base nas coordenadas X, Y e T das pistas. Movimentos celulares em microcanais com constrição foram avaliados manualmente (o número de células que passaram por uma determinada constrição foi contado e o tempo necessário para a passagem da constrição foi calculado de acordo com o número de quadros de imagem que foram tirados até a passagem estar completa). Para alguns experimentos exemplares, Hoechst 33342 (ab228551, Abcam, Cambridge, Reino Unido) foi adicionado ao meio de cultura celular para destacar os núcleos.
2.3 Segmentação de filmes de experimentos de microcanais
A segmentação foi feita com o pacote scikit-image em Python. 24 Primeiro, o ruído foi removido com um filtro mediano e uma imagem de fundo foi calculada tomando o valor do pixel mediano para cada posição em todos os quadros do vídeo. Em seguida, essa imagem de fundo foi subtraída de cada quadro para remover os microcanais da imagem. Um filtro médio foi aplicado para suavizar a imagem novamente, e um filtro top-head preto foi usado para melhorar o sinal das células. Por fim, as células foram detectadas com um limiar de iene, e as operações morfológicas foram usadas para melhorar o resultado.
2.4 Imunofluorescência
As citospinas de todas as linhas celulares foram preparadas e incubadas com um anticorpo monoclonal anti-lamina A/C (1:200; sc-7292, Santa Cruz Biotechnologies, Dallas, EUA). O kit de anticorpos VectaFluor Excel Dylight 594 (Vector Laboratories, Burlingame, CA, EUA) foi usado para detecção. Um microscópio confocal Leica TCS SP8 (Leica Microsystems, Wetzlar, Alemanha) foi usado para imagens 3D. As configurações foram as seguintes: HC PL APO 63x/1,3 GLYC CORR, Cs2; lasers: 405 nm DMOD Compact, Vermelho 594 nm e Verde 488 nm. O tamanho de pixel apresentado foi de 130 nm em cada direção de coordenada. Além disso, o passo z tinha um tamanho de 0,13 mm. 25 , 26
3. RESULTADOS
3.1 Perfis proteômicos globais mostram semelhanças na expressão de proteínas relacionadas à motilidade entre linhagens de células cHL derivadas de células T e ALK − ALCL
Como estávamos interessados em ver se as duas linhagens de células cHL derivadas de células T tinham perfis proteômicos mais semelhantes aos de cHL ou ALCL derivados de células B, os perfis proteômicos globais foram determinados e comparados. Ambas as linhas celulares cHL derivadas de células T, L-540 e HDLM-2, agrupadas com as linhas celulares cHL L-428 e L-1236 no agrupamento global não supervisionado (Figura 1A ). No entanto, em um agrupamento não supervisionado incluindo apenas genes relacionados à motilidade, L-540 e HDLM-2 agruparam-se de perto com as linhas celulares ALK − ALCL MAC-1 e MAC-2A, indicando que a maquinaria intracelular aplicada para movimentos celulares é mais semelhante a a de ALK − ALCL (Figura 1B). As subunidades do complexo de proteína relacionada à actina (Arp)2/3 foram expressas em níveis 1,33 vezes a 1,59 vezes mais altos nas linhagens de células cHL derivadas de células T L-540 e HDLM-2 em comparação com as linhagens de células cHL L -428 e L-1236. MYH9, um componente do complexo miosina II, também apresentou expressão 1,46 vezes maior em L-540 e HDLM-2 em comparação com L-428 e L-1236. CDC42 mostrou expressão 1,63 vezes maior em L-540 e HDLM-2 do que em L-428 e L-1236.
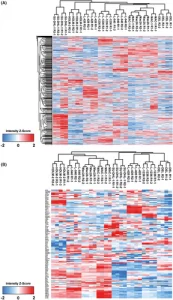
FIGURA 1
Agrupamentos hierárquicos não supervisionados de linhagens celulares ALCL e Hodgkin de acordo com dados de proteômica. (A) Agrupamento hierárquico não supervisionado com base em todos os grupos de proteínas quantificados. (B) Agrupamento hierárquico não supervisionado após o mapeamento de proteínas relacionadas ao movimento celular
3.2 Linhas celulares cHL derivadas de células T compartilham características de movimento de linha de base com linhas celulares cHL e ALK − ALCL
Como já observamos diferenças na migração celular entre ALCL e cHL, 27 agora estávamos interessados em estudar como as linhagens de células cHL derivadas de células T se movem. Observando a velocidade de migração da linha de base, pudemos confirmar em microcanais com largura de 8 µm e altura de 10 µm que as linhas de células cHL L-1236 e L-428 se movem a uma velocidade significativamente menor (0,76 e 0,90 µm/min, respectivamente) do que ALK + (1,52 e 1,45 µm/min) e ALK − ALCL (1,63 e 1,31 µm/min; ANOVA de uma via com pós-teste de Bonferroni para comparações múltiplas; Figura 2 ). As linhas de células cHL derivadas de células T L-540 e HDLM-2 foram ainda mais lentas (ambas 0,41 µm/min) em comparação com as linhas de células cHL L-1236 e L-428; no entanto, essa diferença não foi significativa.
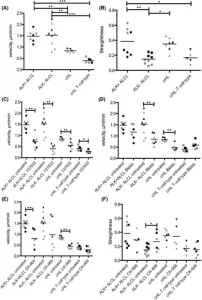
FIGURA 2
Características de movimento de linha de base de linhagens celulares ALCL e Hodgkin e movimento após tratamento com inibidor em microcanais retos com altura de 10 µm e largura de 8 µm. (A) Velocidade baseada em passos de linhagens celulares ALK + , ALK − ALCL e Hodgkin. Pelo menos três experimentos independentes por linhagem celular (*** p < 0,001, ** p < 0,01, ANOVA de uma via com pós-teste de Bonferroni para comparações múltiplas). (B) Retidão das linhas celulares ALK + , ALK − ALCL e Hodgkin. Pelo menos três experimentos independentes por linhagem celular (*** p < 0,001, ** p < 0,01, ANOVA de uma via com pós-teste de Bonferroni para comparações múltiplas). (C). Velocidade baseada em passos de ALK +, ALK − Linhagens celulares ALCL e Hodgkin após tratamento com o inibidor ROCK Y27632 (30 µM). Pelo menos três experimentos independentes por linhagem celular (* p < 0,05, ** p < 0,01, teste de Mann-Whitney). (D) Velocidade baseada em etapas de linhagens celulares ALK + , ALK − ALCL e Hodgkin após tratamento com o inibidor de miosina II Blebbistatin (15 µM). Pelo menos três experimentos independentes por linhagem celular (** p < 0,01, teste de Mann-Whitney). (E) Velocidade baseada em etapas de linhagens celulares ALK + , ALK − ALCL e Hodgkin após tratamento com o inibidor de actina CK-666 (50 µM). Pelo menos três experimentos independentes por linhagem celular (** p< 0,01, teste de Mann-Whitney). (F) Retidão das linhagens celulares ALK + , ALK − ALCL e Hodgkin após tratamento com o inibidor de actina CK-666 (50 µM). Pelo menos três experimentos independentes por linhagem celular (* p < 0,05, teste de Mann-Whitney). Marcado em cinza: ALK + ALCL: DEL, ALK − ALCL: MAC2A, cHL: L-1236, cHL derivado de células T: L-540. Rotulado em preto: ALK + ALCL: SU-DHL-1, ALK − ALCL: MAC1, cHL: L-428, cHL derivado de células T: HDLM-2
Em relação à morfologia das células em movimento, as linhas de células cHL L-1236 e L-428 avançaram lentamente, assemelhando-se ao modo A1 de migração amebóide descrito anteriormente. 28 As células das linhagens de células cHL derivadas de células T apresentaram predominantemente muito pouco movimento, com algumas células com alta velocidade e uma estrutura semelhante a urópode na extremidade traseira da célula, como no modo A2 de migração amebóide descrito anteriormente . O modo A2 nunca foi observado nas linhas de células cHL L-428 e L-1236. Em contraste, as linhagens celulares ALK + ALCL DEL e SU-DHL-1 apresentaram frequentemente células rápidas com morfologia A2. O ALK –As linhagens de células ALCL MAC1 e MAC2A apresentaram alta plasticidade entre os diferentes tipos de migração, com algumas células apresentando morfologia mesenquimal enquanto outras apresentavam morfologia A1 e A2. Em particular, o MAC2A tinha frequentemente células giratórias que giravam para trás e para frente (Figura 2B , Supporting Information Movies S1–S8).
3.3 Os inibidores prejudicam a velocidade das células, mas têm pouco impacto no modo de migração
Em seguida, objetivamos decifrar quais fatores relacionados à migração têm maiores efeitos sobre a motilidade das células tumorais ALCL e cHL. Primeiro, aplicamos o inibidor de ROCK Y27632 (30 µM) e analisamos o efeito na capacidade de migração das células. Todas as linhagens celulares testadas apresentaram comprometimento significativo de sua motilidade após a aplicação de Y27632 (teste de Mann-Whitney), com desaparecimento completo do modo de movimento A2. O efeito foi mais forte nas linhas celulares ALK + e ALK − ALCL, particularmente na linha celular ALK − ALCL MAC2A, enquanto os efeitos nas linhas celulares cHL derivadas de células T e cHL foram menos pronunciados (Figura 2C). A morfologia da migração foi comparável em todas as linhagens celulares tratadas, que mostraram principalmente células com morfologia A1 e saliências da membrana celular, que ocorreram frequentemente na parte traseira da célula, mas também na borda frontal da célula em algumas células (Tabela 1 , Figura 3 , Figuras de Informação de Apoio S1 e S2 e Filmes S9–S11).
TABELA 1. Modo preferencial de movimento observado em células em movimento de acordo com Agarwal et al. 28
| Linfoma | Linha celular | Linha de base | Y27632 | Blebbistatina | CK−666 |
|---|---|---|---|---|---|
| ALK + ALCL | DEL | A1 + A2 | A1 + mesenquimal | A1 + A2 com saliências na parte traseira | A1 + A2, muitas células param |
| SU-DHL-1 | A1 + A2 | A1 com saliências | A1 + A2 com saliências na parte traseira | A1 + A2, muitas células param | |
| ALK – ALCL | MAC1 | A1 + A2 | mesenquimal | A1 + A2 com saliências na parte traseira | A1 + A2 |
| MAC2A | A1 + A2 | A1/sem motilidade | A1 + A2 com saliências na parte traseira | A1 + A2 | |
| cHL | L-428 | A1 | A1 com saliências | A1 com saliências na parte traseira | A1 |
| L-1236 | A1 | A1 com saliências | A1 com saliências na parte traseira | A1 | |
| cHL derivado de células T | HDLM-2 | A1 + A2 | A1 | A1 + A2 + mesenquimal | A1 + A2 |
| L-540 | A1 + A2 | A1 com saliências | A1 + A2 + mesenquimal | A1 + A2 |
Observação
-
Os filmes foram analisados quanto à morfologia das células em movimento. As células imóveis foram negligenciadas nesta análise. A1 e A2 representam formas de movimento amebóide.
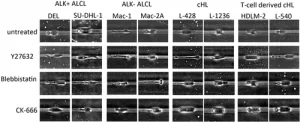
FIGURA 3
Morfologia das células em movimento na linha de base e após o tratamento com inibidor em microcanais retos com altura de 10 µm e largura de 8 µm. As linhas celulares ALK + ALCL DEL e SU-DHL-1, ALK − ALCL linhas celulares MAC1 e MAC2A, linhas celulares cHL L-428 e L-1236 e as linhas celulares cHL derivadas de células T L-540 e HDLM-2 são exibido. Primeira linha, as células são mostradas na linha de base, segunda linha após inibição de ROCK com 30 µM de Y27632, terceira linha após inibição de miosina II com 15 µM de Blebbistatina, última linha após inibição do complexo Arp2/3 de proteína relacionada à actina com 50 µM de CK-666 é mostrada . Exemplos representativos em microscopia de contraste de fase com ampliação de 10x foram escolhidos
Em seguida, testamos o efeito do inibidor de miosina II blebbistatina (15 µM) em todas as linhagens celulares. Diferenças significativas na velocidade foram observadas aqui entre as linhagens celulares ALK − ALCL e as linhagens celulares cHL. Surpreendentemente, as linhagens de células cHL derivadas de células T podem até se mover um pouco melhor após a aplicação de blebistatina (Figura 2D ), sugerindo que essas linhagens de células ainda podem ser capazes de se mover após a inibição da miosina II em relação aos seus níveis mais elevados de miosina II endógena, conforme observado na triagem proteômica global. Após a aplicação de blebistatina, a morfologia das células migratórias mudou regularmente, pois as células apresentavam problemas com a retração de sua parte traseira, levando a caudas longas em suas extremidades traseiras (Figura 3 )., Informações de suporte Figura S3 e Filmes S12–S14). No entanto, células que se movem no modo A2 foram ocasionalmente encontradas, às vezes com uma cauda longa no urópode (Figura 3 , MAC1 e L-540).
Após a inibição do complexo Arp2/3 utilizando o inibidor CK-666 (50 µM), todas as linhagens celulares apresentaram motilidade celular prejudicada, o que foi significativo para as linhagens celulares ALK + ALCL e cHL. Considerando que os tratamentos com Y27632 e blebbistatina não tiveram efeitos na retilineidade das células, surpreendentemente, as linhagens celulares ALK − ALCL MAC1 e MAC2A apresentaram um aumento significativo na retilineidade após a administração de CK-666, o que provavelmente foi relacionado à baixa velocidade global, com algumas células com morfologia A2 movendo-se em linha reta a uma velocidade relativamente alta. A morfologia das células tratadas com CK-666 foi pouco alterada em comparação com as células não tratadas (Figura 3 ).
3.4 ALK + ALCL são superiores ao cHL na superação de constrições em microcanais devido a lâminas nucleares mais flexíveis
Como também estávamos interessados em elucidar como as diferentes linhagens celulares de linfoma lidariam com espaços severamente confinados semelhantes à passagem de células tumorais entre células endoteliais, a migração em microcanais com constrições de 12 × 4 µm foi monitorada. Enquanto a maioria das células ALK + ALCL podem passar por várias constrições com uma velocidade relativamente alta (Figura 4 ), as linhas celulares cHL, incluindo as linhas celulares cHL derivadas de células T, foram menos eficientes na passagem de constrições. A maioria das células das linhagens de células cHL derivadas de células T e cHL passaram muito tempo na frente ou dentro da constrição em comparação com as linhas celulares ALK + ALCL. O tempo necessário para passar a primeira constrição foi significativamente maior para linhagens de células cHL derivadas de células T em comparação com ALK+ ALCL ( p < 0,05, teste de Kruskal-Wallis com pós-teste de Dunn para comparações múltiplas; Figura 4 ). Mais uma vez, este fato aponta para linhas celulares cHL derivadas de células T ocupando uma posição intermediária entre as linhas celulares cHL derivadas de células ALCL e B.
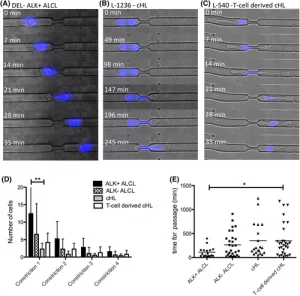
FIGURA 4
Movimento de linhagens celulares Hodgkin e ALCL em microcanais com 12 µm de diâmetro e 4 µm de constrição. (A) Passagem da primeira constrição por uma célula DEL (ALK + ALCL) em 28 min. O núcleo é destacado em azul (corante Hoechst), aumento de 40x. (B) Passagem da primeira constrição por uma célula L-1236 (cHL) dentro de 266 min. O núcleo é destacado em azul (corante Hoechst), aumento de 40x. (C) Passagem da primeira constrição por uma célula L-540 (cHL derivada de células T) dentro de 35 min. O núcleo é destacado em azul (corante Hoechst), aumento de 40x. (D) Número de células que passam pelas primeiras quatro constrições de acordo com o número de constrições e o tipo de linfoma. ** p< 0,01, teste de Kruskal-Wallis com pós-teste de Dunn para comparações múltiplas. 4-5 réplicas para cada linha celular, duas linhas celulares por entidade de linfoma. (E) Tempo (min) necessário para a passagem da primeira constrição. Cada ponto representa uma célula. Duas linhas celulares por entidade de linfoma. * p < 0,01, teste de Kruskal-Wallis com pós-teste de Dunn para comparações múltiplas
A passagem através de constrições pode ser limitada devido à rigidez nuclear, em que a lâmina A (e sua variante de emenda da lâmina C) contribui para a rigidez da lâmina nuclear, enquanto a lâmina B1 e B2 contribuem para uma lâmina nuclear mais flexível. Portanto, verificamos os dados de proteômica para a expressão da lamina A e B1/B2. As linhas celulares ALK + ALCL tiveram a menor expressão de proteína lamina A (valor médio de intensidade 158.257.750 em ALK + ALCL vs valores médios de intensidade 240.863.250, 244.062.500 e 260.886.750 em ALK − ALCL, cHL e cHL derivado de células T, respectivamente). Em contraste, ALK + ALCL teve a maior expressão de lamina B1 (valor médio de intensidade 219.675.000 vs valores médios de intensidade 142.970.750, 152.759.000 e 183.119.500 em ALK −ALCL, cHL e cHL derivado de células T, respectivamente), sugerindo núcleos relativamente macios e flexíveis em ALK + ALCL (Figura 5 ) em comparação com as outras linhagens celulares. Todas as linhagens de células testadas também foram investigadas por imunofluorescência para expressão da proteína lamina A/C. Aqui, observamos a expressão mais forte de lamin A/C em linhas celulares cHL e derivadas de células T, com uma distribuição um pouco mais heterogênea da expressão de lamin A/C em linhas celulares ALK − ALCL e uma expressão fraca e heterogênea em ALK + Linhas celulares ALCL (Figura 5 ). Assim, a expressão da lamina A/C foi inversamente correlacionada com a capacidade das linhas celulares de passar por constrições.
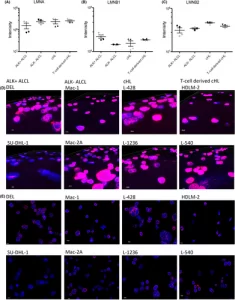
FIGURA 5
Expressão da lamina A/C nas lâminas nucleares das linhas celulares Hodgkin e ALCL. (A) Expressão da proteína Lamin A em linhagens celulares cHL e ALCL de acordo com dados de proteômica global. (B) Expressão da proteína Lamin B1 em linhagens celulares cHL e ALCL de acordo com dados de proteômica global. (C) Expressão da proteína Lamin B2 em linhagens celulares cHL e ALCL de acordo com dados de proteômica global. Marcado em cinza: ALK + ALCL: DEL, ALK − ALCL: MAC2A, cHL: L-1236, cHL derivado de células T: L-540. Rotulado em preto: ALK + ALCL: SU-DHL-1, ALK −ALCL: MAC1, cHL: L-428, cHL derivado de células T: HDLM-2. (D) imagem confocal 3D de núcleos celulares em imunofluorescência Lamin A/C, núcleos contrastados com DAPI, ampliação de 63x, linhas celulares conforme indicado. (F) Corte de corte através de núcleos celulares em imunofluorescência Lamin A/C, núcleos contrastados com DAPI, ampliação de 63 ×, linhas celulares conforme indicado
4. DISCUSSÃO
Até onde sabemos, este estudo é o primeiro a avaliar a motilidade celular para classificar diferentes linhagens celulares de linfoma. Pudemos demonstrar em estudos anteriores 19 , 27 que entidades de linfoma com semelhanças em morfologia, imuno-histoquímica e características moleculares apresentam diferenças em suas propriedades migratórias. Embora cHL com rearranjos clonais de TCR tenham sido descritos, 15devido à raridade de tais casos, sua existência era duvidosa e não estava claro como melhor classificá-los. Aqui, apresentamos dados que mostram que a morfologia das células em movimento de linhagens de células cHL derivadas de células T tem semelhanças com ALCL, que provavelmente se relaciona predominantemente com a origem das células T das células. No entanto, a baixa capacidade das células HRS derivadas de células T de se moverem tem uma maior semelhança com cHL. As características morfológicas das células HRS – enormes, pleomórficas, às vezes binucleadas – correlacionam-se bem com a incapacidade de se mover, o que também pudemos demonstrar para cHL em amostras de tecido primário. 19 A incapacidade de se mover de linhagens de células cHL derivadas de células T e B combina com a observação clínica de que PAX5 primário −Casos de cHL expressando moléculas citotóxicas (e, portanto, provavelmente derivadas de células T) não diferiram em estágio ou LDH de outros casos de cHL, 12 enquanto os linfomas periféricos de células T apresentaram doença em estágio avançado significativamente mais frequentemente. A incapacidade das células HRS de se propagarem de um linfonodo para outro é refletida na frequente doença em estágio limitado e no envolvimento contíguo dos linfonodos.
A raridade desses casos de linfoma também foi a razão pela qual não tivemos a chance de estudar suspensões desses casos primários em microcanais. Embora tenhamos testado suspensões de cHL primário convencional e ALCL, o número de células tumorais CD30+ que entram nos microcanais era muito baixo para tirar conclusões significativas. A maioria das células em movimento aqui representava células T reativas do microambiente. Portanto, atualmente devemos confiar nas linhagens de células cHL derivadas de células T como um bom modelo para esta doença.
As linhagens de células cHL derivadas de células T apresentaram algumas células móveis com morfologia urópode e amebóide típica A2, que também observamos em linhagens de células ALCL. A formação de um urópode com agrupamento de TCRs em uma sinapse imunológica dinâmica, também chamada de quinapse, 29 é frequentemente observada para células T normais. 30 Portanto, o modo de movimento das linhagens de células cHL derivadas de células T reflete sua origem de células T. No entanto, a motilidade geralmente fortemente prejudicada em linhagens de células cHL derivadas de células T assemelha-se a cHL derivada de células B.
No que diz respeito à suscetibilidade a diferentes inibidores, o inibidor ROCK Y27632 teve algum efeito nas linhagens de células cHL derivadas de células T. Como observado em quase todas as linhagens celulares, foram observadas caudas longas na parte traseira da célula, como também descrito anteriormente no contexto da retração da cauda dos monócitos após o bloqueio de Rhoa . 31 Enquanto as linhagens celulares ALCL apresentaram uma motilidade fortemente diminuída após a aplicação da maioria dos inibidores, os efeitos foram menos pronunciados em linhagens celulares cHL e quase anulados em linhagens celulares cHL derivadas de células T, indicando que as células cHL derivadas de células T já têm máquinas de movimento prejudicadas na linha de base. Para ALK +ALCL, a presença do gene de fusão NPM-ALK foi descrita para regular positivamente o movimento das células pela fosforilação de VAV1 e VAV3 e consequente ativação de RAC1. 32 , 33 Além disso, foi demonstrado que o gene de fusão NPM-ALK contribui para a polarização das células e substitui a estimulação do TCR. No entanto, em nossos dados, as linhagens celulares ALK + ALCL se comportaram de forma semelhante a ALK − ALCL em sua migração, embora os componentes do citoesqueleto ativados possam diferir, como também observamos através da abordagem proteômica global. Ambos ALK + e ALK −As linhas celulares ALCL foram eficientes na passagem de constrições de 4 µm, particularmente para a primeira constrição da série. A mecânica e a rigidez da membrana nuclear das células do linfoma podem levar à ruptura nuclear e danos ao DNA durante a passagem por uma constrição, 34 , 35 resultando em apoptose. Em consonância com a incapacidade das linhagens de células cHL de passar por constrições, observamos forte expressão de lâmina A/C na lâmina nuclear. A segmentação aumentada dos núcleos pela lamina A/C foi previamente observada em células tumorais de cHL. 36
Com base na baixa eficiência do movimento, as linhagens de células cHL derivadas de células T são diferentes de outras linhas de células de linfoma de células T CD30+. No perfil de expressão gênica, as linhas de células cHL derivadas de células T assemelhavam-se muito às linhas de células cHL convencionais. 18 No entanto, o resultado clínico de PAX5 − cHL após o tratamento de cHL foi inferior ao do cHL convencional, 12 sugerindo que existem diferenças entre cHL derivado de células B e células T. Estudos comparando o comportamento clínico do cHL derivado de células T com o ALCL convencional são escassos até o momento devido à raridade desses casos.
Em resumo, nossos dados derivados de linhagens celulares sugerem que casos com a morfologia de cHL e origem de células T das células neoplásicas (cHL derivado de células T) provavelmente não devem ser mesclados com cHL nem com ALCL. É necessária uma caracterização aprofundada do comportamento clínico de um maior número desses casos.
CONTRIBUIÇÃO DO AUTOR
Julia Bein: curadoria de dados (liderança); Análise formal (igual); Investigação (igual). Nadine Flinner: Metodologia (igual); Recursos (iguais); Software (líder). Björn Häupl: Curadoria de dados (igual); Análise formal (igual); Visualização (igual). Aastha Mathur: Curadoria de dados (igual); Análise formal (igual). Olga Schneider: Curadoria de dados (igual); Análise formal (igual). Marwa Abu-Ayyad: Software (igual). Martin-Leo Hansmann: Conceituação (igual); Supervisão (igual). Matthieu Piel: Conceituação (igual); Metodologia (liderança); Administração do projeto (igual); Recursos (iguais); Supervisão (igual). Thomas Oellerich:Curadoria de dados (igual); Recursos (iguais); Supervisão (igual). Sylvia Hartmann: Conceituação (liderança); Curadoria de dados (igual); Análise formal (igual); Aquisição de financiamento (apoio); Investigação (igual); Administração do projeto (igual); Supervisão (líder); Visualização (lead); Redação – rascunho original (liderança).
RECONHECIMENTOS
Este projeto foi apoiado pela Deutsche Forschungsgemeinschaft (subvenção HA6145/6-1). O NF é financiado pelo Mildred Scheel Career Centre Frankfurt (Deutsche Krebshilfe). Agradecemos ao Prof. Ralf Küppers e Kerstin Heise, University Duisburg-Essen, Alemanha, pelo perfil de STR e verificação de linhagens celulares. Agradecemos ao Prof. Olaf Merkel, Universidade Médica de Viena, Áustria, por fornecer as linhas celulares MAC-1 e MAC-2A. Agradecemos a Yvonne Steiner pela excelente imagem confocal a laser 3D. Financiamento de acesso aberto habilitado e organizado pelo Project DEAL.
CONFLITO DE INTERESSES
Os autores não relatam nenhum conflito de interesse.





